DICOM파일 다루기 with python
DICOM 파일 다루기
pydicom
최근 X-ray 의료 데이터를 다루를 캐글 대회에 참여하게 되었습니다.
처음 해보는 분야이고 매번 해봐야지 해봐야지.. 생각만 하다가 직접 맞딱드리니 어려움이 이만 저만이 아니였습니다.
https://www.kaggle.com/c/siim-covid19-detection
의료 데이터는 DICOM 또는 NIfTI라는 포맷으로 저장되어 있습니다.
SIIM 대회에서는 DICOM 파일로 데이터를 주었기 때문에 DICOM파일에 관련한 내용을 다루고자 합니다.
파이썬에서는 pydicom이라는 라이브러리로 다룰 수 있습니다.
pydicom은 DICOM 파일 작업을위한 순수 Python 패키지입니다. 쉬운 “pythonic”방식으로 DICOM 데이터를 읽고, 수정하고, 쓸 수 있습니다. -pydicom 공식 깃허브
Installation
Using pip:
pip install pydicom
Using conda:
conda install -c conda-forge pydicom
그 외 설치 가이드는 여기를 눌러주세요
Example
```python import-lib import pydicom from pydicom.pixel_data_handlers.util import apply_voi_lut import cv2 from glob import glob import matplotlib.pyplot as plt
위 명령어를 통해 library들을 불러옵니다
dataset_dir = ‘../input/siim-covid19-detection’ dicom_paths = glob(f’{dataset_dir}/train///*.dcm’) dicom_paths[0:10]
데이터 셋 전체에서 10개의 경로를 glob 함수를 통해 출력 해보겠습니다.
[’../input/siim-covid19-detection/train/cd5dd5e6f3f5/b2ee36aa2df5/d8ba599611e5.dcm’, ‘../input/siim-covid19-detection/train/49358afcfb80/60a49211f5df/29b23a11d1e4.dcm’, ‘../input/siim-covid19-detection/train/e4b50e7402c3/59f646771321/8174f49500a5.dcm’, ‘../input/siim-covid19-detection/train/e4b50e7402c3/d289a11b2e85/d54f6204b044.dcm’, ‘../input/siim-covid19-detection/train/92aad2d01be8/60fe0c912619/d51cadde8626.dcm’, ‘../input/siim-covid19-detection/train/b524c80e729a/2702319e5a1d/47d014f9055a.dcm’, ‘../input/siim-covid19-detection/train/3927f0e451cc/d5d5b1c75389/89fd7f185d77.dcm’, ‘../input/siim-covid19-detection/train/ba27bcbd6881/09fb8b5e1ce8/7c40e04c6163.dcm’, ‘../input/siim-covid19-detection/train/ba27bcbd6881/d300cb010b90/6a93346150a4.dcm’, ‘../input/siim-covid19-detection/train/e38bf848a4b2/e16ffae4da8c/5b687c54d3fd.dcm’]
## 전처리
```python
dicom= pydicom.read_file(dicom_paths[0])
data = apply_voi_lut(dicom.pixel_array, dicom)
data
if dicom.PhotometricInterpretation == 'MONOCHROME1':
data = np.amax(data) - data # amax array 의 최댓값을 반환하는 함수
data= data - np.min(data)
data= data / np.max(data)
data= (data*255).astype(np.uint8)
* dicom에 저는 dicom_paths중 0번째 값을 넣어줬습니다
VOI LUT (DICOM 장치에서 사용 가능한 경우)는 원시 DICOM 데이터를 “인간 친화적인”보기로 변환하는 데 사용됩니다.
사용하지 않겠다 하신다면 dicom.pixel_array를 바로 하시면 됩니다.
dicom.PhotometricInterpretation
-> 'MONOCHROME1'
dicom.PhotometricInterpretation에 관하여
Photometric Interpretation은 dicom파일의 광도를 해석해줍니다.
Output 값으로
우리가 궁금한건 여기서 MONOCHOME1입니다
MONOCHOME1이란?
픽셀 데이터는 단일 단색 이미지 평면을 나타냅니다. 최소 샘플 값은 VOI 그레이 스케일 변환이 수행 된 후 흰색으로 표시됩니다. 이 값은 픽셀 당 샘플 (0028,0002)의 값이 1 인 경우에만 사용할 수 있습니다. 네이티브 (비 압축) 또는 캡슐화 (압축) 형식의 픽셀 데이터에 사용할 수 있습니다.
결국은 X-ray 이미지를 사용하고 있으니 단색 이미지 평면인 흑백이라고 알려주는 것과 같습니다.
더 자세한 내용은 여기
위 코드에서 마지막으로 normalization을 마치고 나면 아래와 같은 결과를 얻을 수 있습니다.
#output
[[ 24 24 21 ... 127 125 128]
[ 20 17 17 ... 125 125 128]
[ 26 22 18 ... 122 123 125]
...
[ 46 40 42 ... 20 0 0]
[ 43 41 45 ... 20 0 0]
[ 37 40 41 ... 12 0 0]]
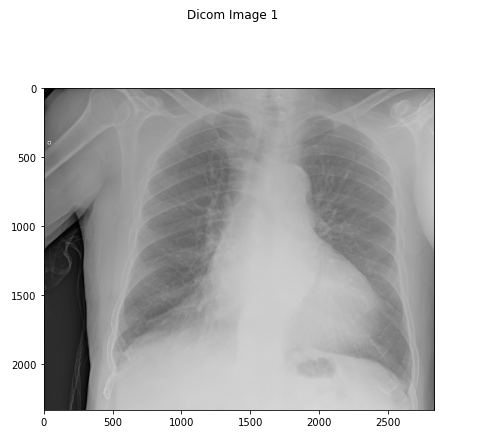
시각화
def plot_img(img, size=(7,7), is_rgb=True, title="Dicom Image 1", cmap='gray'):
plt.figure(figsize=size)
plt.imshow(img, cmap=cmap)
plt.suptitle(title)
plt.show()
plot_img(data)
오늘은 dicom파일을 다루는 기초적인 방법을 알아봤습니다.
이 내용이 읽으시는 분들에게 유익했으면 좋겠습니다.
출처: http://dicom.nema.org/medical/Dicom/2018d/output/chtml/part03/sect_C.11.2.html https://dicom.innolitics.com/ciods/ct-image/image-pixel/00280004